Error in lda(x, y, reduced_rank, k) : rank out of bounds
【报错原因】 数据问题 【解决方法】 更换响应变量
运算因错误中止,原因:Error in data.frame(…, check.names = FALSE) :参数值意味着不同的行数: 546, 528
【报错原因】 因变量和自变量缺失数目不同,预测值和真值数目不匹配 【解决方法】 插补空值或者删除空值
Error in $<-.data.frame(*tmp*, "mfit", value = c(1 = -11.7386871143257, :替换数据里有319行,但数据有423请检查表格变量类型是否符需要转换,变量值是否需要插补空值,变量名是否存在非法字符!
【报错原因】 变量里含有空值 【解决方法】 建议将变量的空缺值进行插补
Error in names(x) <- value :'names' attribute [4] must be the same length as the vector [0]
【报错原因】 自变量没填 【解决方法】 填写自变量
Error in qgrubbs(q, n, type, rev = TRUE) : n must be in range 3-30
【报错原因】 检验方法为20时,样本量必须在3-30之间 【解决方法】 控制样本量为3-30
Error: Cochran-Armitage test for trend must be used with rx2-table
【报错原因】 选择变量出现问题,不是二分类 【解决方法】 需要确保数据是一个两列的列联表,分别代表二分类响应
Error in solve.default(SKK) :system is computationally singular: reciprocal condition number = 2.16588e-21
【报错原因】 数据问题算不出 【解决方法】 更改协整成分
Error in lrtest.default(model_one, model_two) :’list’ object cannot be coerced to type ‘double’请检查表格变量类型是否符需要转换,变量值是否需要插补空值,变量名是否存在非法字符!
【报错原因】 上游节点为调查设计回归分析时,如果遇到该错误,需要重启软件。 【解决方法】 设置好参数后重启软件,直接运行似然比检验节点
Error in dimnames(x) <- dn :length of 'dimnames' [1] not equal to array extent
【报错原因】 变量存在空值 【解决方法】 插补空值
Error in binom.test(sum(mydata1[, var_input[i]] < mydata2[, var_input[i]]), :'n'必需是大于等于'x'的正整数
【报错原因】 需要配对分组变量 【解决方法】 建议更换分组变量
Error in getActiveRowSpan(estimates): Could not identify rows with actual data
【报错原因】 通常为节点运算结果绘制森林图的表格一些变量存在NA,通常为某些变量的P值存在NA 【解决方法】 在计算的时候剔除该变量
Error in imputeTS::na_ma(v, k = k, weighting = “linear”) :At least 2 non-NA data points required in the time series to apply na_ma.
【报错原因】 ID id筛选之后数据没了 【解决方法】 更换数据或变量
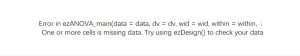
Error in ezANOVA_main(data = data, dv = dv, wid = wid, within = within, :One or more cells is missing data. Try using ezDesign() to check your data
【报错原因】 条件变量和ID变量没有配对,不是重复测量数据 【解决方法】 建议更换变量,使得每个重复数量相等
Error in quantile.default(WD[, var_input[i]], 0.25) :’na.rm’如果设为FALSE的话不允许有遺漏值和NaN
【报错原因】 变量存在空值 【解决方法】 插补空值或者删除空值
Error in solve.default(denom, numr) :system is computationally singular: reciprocal condition number = 5.26589e-23
【报错原因】 卡方计算出现问题,两个变量选择不对 【解决方法】 建议更换变量
Error in rmst2(subWD[, var_time], subWD[, var_dependent], subWD[, var_independent], :object ‘NOTE’ not found
【报错原因】 生存状态和ARM变量选择有误 【解决方法】 检查并重新选择生存状态和ARM变量
分析单元“8.分位数节点”运算出错,请检查。Error in rcspline.eval(x, knots = knots, inclx = TRUE, pc = pc, fractied = fractied) :
【报错原因】 输入分位数 数量要大于2;数据不能过于集中,要有明显的四分位区间 【解决方法】 输入大于等于3的分位数;更换数据
Error in num point[[ind pred]]: attempt to select less thanone element in get1index.
【报错原因】 节点数不支持计算 【解决方法】 更改节点数












![Error in names(x) <- value :'names' attribute [4] must be the same length as the vector [0]-决策链社区论坛](https://bbs.statsape.com/wp-content/uploads/2025/04/20250416114709771-105-300x53.jpg)




![Error in dimnames(x) <- dn :length of 'dimnames' [1] not equal to array extent-决策链社区论坛](https://bbs.statsape.com/wp-content/uploads/2025/04/20250415134026868-59-300x54.jpg)

![Error in binom.test(sum(mydata1[, var_input[i]] < mydata2[, var_input[i]]), :'n'必需是大于等于'x'的正整数-决策链社区论坛](https://bbs.statsape.com/wp-content/uploads/2025/04/20250414152122184-16-300x54.jpg)




![Error in quantile.default(WD[, var_input[i]], 0.25) :'na.rm'如果设为FALSE的话不允许有遺漏值和NaN-决策链社区论坛](https://bbs.statsape.com/wp-content/uploads/2025/04/20250415160044471-62-300x52.jpg)


![Error in rmst2(subWD[, var_time], subWD[, var_dependent], subWD[, var_independent], :object 'NOTE' not found-决策链社区论坛](https://bbs.statsape.com/wp-content/uploads/2025/04/20250414123017750-10-300x53.jpg)

![Error in num point[[ind pred]]: attempt to select less thanone element in get1index.-决策链社区论坛](https://bbs.statsape.com/wp-content/uploads/2025/04/20250416135411726-122-300x52.jpg)

